1. Introduzione e finalità dello studio
I formaggi a pasta dura devono le loro caratteristiche chimico-fisiche quali e quantitative a fattori fondamentali quali: la qualità del latte crudo, le condizioni di allevamento delle bovine produttrici e le tecnologie di trasformazione. Un complesso insieme di specie microbiche di variabile origine e composizione ha tuttavia una sensibile influenza sulla la composizione microbica del prodotto maturato e come per qualsiasi alimento soggetto a fenomeni di natura fermentativa, la composizione del microbiota gioca un ruolo vitale nella trasformazione e maturazione del formaggio. I microrganismi presenti nel latte d’origine, insieme a quelli dei fermenti e dei contaminanti ambientali, condizionano i fenomeni biochimici che influenzano le caratteristiche del formaggio che finiscono per definirne la qualità. Il complesso microbiota del formaggio, e la sua attività enzimatica, guidano e modellano lungo il periodo destinato alla maturazione i processi enzimatici proteolitici, lipolitici e fermentativi che costituiscono il fenomeno definito globalmente come “maturazione” (da 7 a 24 mesi). La letteratura scientifica sui formaggi a pasta dura e simili è soggetta a continuo arricchimento, e in particolare negli ultimi tempi è supportata da tecniche di indagine molecolare d’avanguardia. Studi recenti si concentrano sull’attività di microrganismi utili (come i batteri lattici, LAB) riconoscendo comunque a quelli indesiderati (come i contaminanti clostridiali) una funzione non trascurabile.
La metagenomica si definisce come la tecnica che permette lo studio delle sequenze di DNA di comunità microbiche complesse a partire direttamente da campioni ambientali, senza necessità di impiego di lunghi tempi al fine di isolare e moltiplicare singoli microrganismi.
Questo approccio è particolarmente utile nell’analisi dei prodotti alimentari, inclusi i formaggi, per una serie di motivi:
- Possibilità di identificazione con alta efficienza dei microrganismi. La metagenomica consente di identificare in un campione alimentare tutti i microrganismi presenti, compresi quelli di difficile coltivazione in laboratorio. La tecnica è fondamentale per accedere alla conoscenza della diversità microbica e delle specifiche dinamiche delle comunità microbiche che influenzano i processi di maturazione dei formaggi.
- Realizzare il monitoraggio della Qualità. L’analisi metagenomica consente di monitorare la qualità dei prodotti alimentari durante tutto il processo di produzione e maturazione. Ciò permette anche l’identificazione di batteri nocivi e la loro gestione al fine di migliorare la shelf-life e sicurezza degli alimenti.
- Consentire la conoscenza delle possibili interazioni microbiche. La metagenomica permette di comprendere le possibili interazioni che intervengono tra i diversi microrganismi presenti nei formaggi. Lo studio di queste interazioni è fondamentale per la comprensione dei processi di fermentazione e maturazione, in quanto sono tali processi a influenzae le caratteristiche nutrizionali, sensoriali e funzionali del formaggio.
- Intervenire nelle tecnologie di innovazione e ottimizzazione di prodotto. Le informazioni ottenute attraverso la metagenomica possono essere utilizzate per migliorare e innovare i processi di produzione nel settore caseario. Attraverso l’identificazione e la selezione di specifici ceppi microbici, è possibile ottimizzare la qualità e le proprietà fisiche, chimiche e sensoriali dei prodotti di trasformazione nel dairy.
Inoltre, l’associazione di metodi metagenomici con aggiornati profili metabolici produce potenzialità analitica utile a dirimere problematiche complesse proprio in tema di maturazione dei formaggi. Trattasi in tal caso di “analisi combinata metagenomica-metabolomica”.
2. Metodologia
In questa nota si esplicita con un esempio, riferito a quattro aliquote di formaggio Gran Moravia, corrispondenti a quattro stadi di maturazione usate per l’estrazione del DNA genomico, un’applicazione di analisi metagenomica.
Le quattro aliquote corrispondono a distinte matrici interne di formaggio prodotto a Litovel e con stagionatura al momento del taglio-forma di ca 7 mesi, 13 mesi, 15 mesi e 21 mesi.
L’inizio del periodo di maturazione per le quattro distinte aliquote è quello qui indicato:
19-23.05.23 con taglio forma il 30.11.2023 e prodotto 01-05.05.23
19-2511.22 con taglio forma il 30.11.2023 e prodotto 01-07.11.22
19-25.09.22 con taglio forma il 30.11.2023 e prodotto 01-07.09.22
19-29.03.22 con taglio forma il 30.11.2023 e prodotto 01-29.03.22
Il riferimento ai lotti di produzione è il seguente: CB, SE, OE, NE nell’ordine di 7, 13, 15, 21 mesi.
Le librerie di sequenziamento sono state prodotte su una piattaforma “Illumina” presso i laboratori Macrogen (Corea del Sud).
I dati di sequenza grezzi sono stati sottoposti a controllo di qualità seguito da analisi bioinformatica secondo lo schema di Figura 1.
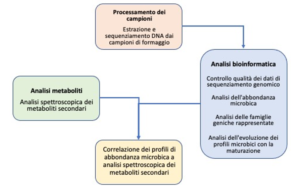
Figura 1. Schema analitico dai campioni di formaggio alla generazione di dati metagenomici.
In particolare, l’analisi bioinformatica ha permesso di studiare l’abbondanza e la diversità delle specie microbiche unitamente alle vie metaboliche e alle famiglie di geni più rappresentate.
La complessità dei dati generati e dell’attività di data mining corrispondente può essere rappresentata solo parzialmente in modo grafico: a tal fine si può far riferimento, per esempio, all’istogramma dei profili tassonomici e ancor più all’abbondanza microbica relativa (Figure 2-1, 2-2, 2-3 e 2-4).
Figura 2-1.
Figura 2-2.
Figura 2-3.
Figura 2-4.
Di particolare importanza è la marcata riduzione dei generi Staphylococcus ed Escherichia che, pur essendo presenti nella prima fase di maturazione, riducono costantemente la loro concentrazione fino alla completa scomparsa nella terza e quarta fase. Al contrario, i generi Lacticasebacillus e Lactobacillus aumentano progressivamente la loro presenza nelle fasi avanzate di maturazione.
L’analisi funzionale ha consentito di stilare un elenco delle famiglie geniche più abbondanti lungo le fasi di maturazione (Figura 3), mentre un’analisi simile è stata effettuata per evidenziare le vie metaboliche più rappresentate durante le fasi di maturazione (Figura 4).
Le vie evidenziate nell’analisi possono essere correlate con le specie microbiche più rappresentate.

Figura 3. Profili funzionali per le 20 famiglie geniche più abbondanti.

Figura 4. Profili funzionali delle 20 vie più abbondanti.
Bibliografia
- Gobbetti, M.; Fox, P.; Neviani, E. (Eds.) The most traditional and popular Italian cheeses. In The Cheeses of Italy: Science and Technology; Springer: Cham, Switzerland, 2018; pp. 99–267.
- Montel, M.C.; Buchin, S.; Mallet, A.; Delbes-Paus, C.; Vuitton, D.A.; Desmasures, N.; Berthier, F. Traditional cheeses: Rich and diverse microbiota with associated benefits. Int. J. Food Microbiol. 2014, 177, 136–154.
- Gatti, M.; Bottari, B.; Lazzi, C.; Neviani, E.; Mucchetti, G. Invited review: Microbial evolution in raw-milk, long-ripened cheeses produced using undefined natural whey starters. J. Dairy Sci. 2014, 97, 573–591.
- Neviani, E.; Bottari, B.; Lazzi, C.; Gatti, M. New developments in the study of the microbiota of raw-milk, long-ripened cheeses by molecular methods: The case of Grana Padano and Parmigiano Reggiano. Front. Microbiol. 2013, 4, 36.
- Quigley, L.; O’Sullivan, O.; Beresford, T.P.; Ross, R.P.; Fitzgerald, G.F.; Cotter, P.D. Molecular approaches to analyzing the microbial composition of raw milk and raw milk cheese. Int. J. Food Microbiol. 2011, 150, 81–94.
- Ercolini, D. Secrets of the cheese microbiome. Nature Foods 2020, 1, 466–467.
- Buchfink, B., Xie, C. & Huson, D. H. Fast and sensitive protein alignment using DIAMOND. Nature Methods. 2015, 12, 59–60.
- Caspi, R., et al. “The MetaCyc database of metabolic pathways and enzymes-a 2019 update.” Nucleic acids research. 2020, 48, 445-453.

Fioritura del glicine – Lab. FERL – Di.S.A.A. – UniMi

